DNT-nin bağlanma sahəsi
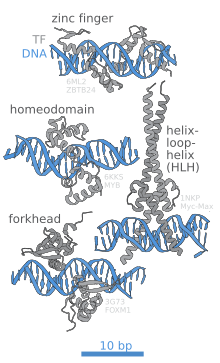
DNT-nin bağlanma sahəsi — DNT bağlanma sahələri, digər molekulların bağlana biləcəyi DNT-də olan bir növ bağlanma sahəsidir. DNT bağlanma sahələri digər bağlanma sahələrindən DNT ardıcıllığının bir parçası olması (məsələn, genom) və DNT bağlama zülalları ilə əlaqəli olmaları ilə fərqlənir[1] . DNT bağlanma yerləri tez-tez transkripsiya faktorları kimi tanınan ixtisaslaşmış zülallarla əlaqələndirilir və bu səbəbdən transkripsiyanın tənzimlənməsi ilə əlaqələndirilir[2] . Müəyyən bir transkripsiya faktoru üçün DNT bağlanma sahələrinin cəminə onun sistromu deyilir. DNT-yə bağlanma sahələri, məhdudlaşdırıcı fermentlər, sahəyə spesifik rekombinazlar və metiltransferazlar kimi digər proteinlərin hədəflərini də əhatə edir[3]. Beləliklə, DNT-yə bağlanma sahələri, bir və ya daha çox DNT bağlayan zülal və ya protein kompleksi ilə xüsusi olaraq bağlanan qısa DNT ardıcıllığı olaraq təyin edilə bilər. Bəzi bağlayıcı sahələrin sürətli təkamül dəyişikliklərinə məruz qalma potensialına sahib olduğu bildirildi[4].
DNT bağlanma sahələrinin növləri[redaktə | mənbəni redaktə et]
DNT-yə bağlanma sahələri bioloji funksiyalarına görə təsnif edilə bilər[5]. Beləliklə, transkripsiya faktorları, məhdudlaşdırma sahələri və rekombinasiya sahələri üçün bağlanma sahələrini ayırd edə bilərik[6][7] . Bəzi müəlliflər bağlayıcı saytların ən uyğun təqdimat üsuluna görə də təsnif edilə biləcəyini irəli sürmüşlər . Bir tərəfdən, məhdudlaşdırma yerləri ümumiyyətlə konsensus ardıcıllığı ilə təmsil edilə bilər[8]. Bunun səbəbi, əksərən eyni ardıcıllıqları hədəf alması və daha az oxşar ardıcıllıqlar üçün məhdudlaşdırma səmərəliliyi dramatik şəkildə azalmasıdır[8]. Digər tərəfdən, müəyyən bir transkripsiya faktoru üçün DNT-yə bağlanma sahələri, ümumiyyətlə fərqli bağlanma sahələri üçün transkripsiya faktorunun yaxınlıq dərəcələri ilə fərqli olur[9]. Bu, konsensus ardıcıllıqlarını istifadə edərək transkripsiya faktoru bağlama yerlərini dəqiq bir şəkildə təmsil etməyi çətinləşdirir və bunlar ümumiyyətlə ardıcıllıq loqotiplərindən istifadə edilərək tez-tez qrafiki şəkildə təsvir olunan mövqeyə aid tezlik matrisləri (PSFM) istifadə olunur[10]. Ancaq bu arqument bir qədər özbaşına olur. Transkripsiya faktorları kimi məhdudlaşdırıcı fermentlər, fərqli yerlərə tədricən, kəskin də olsa yaxınlıq verir və bu səbəbdən PSFM-lər də ən yaxşı şəkildə təmsil olunur. Eynilə, sahəyə məxsus rekombinazlar da fərqli hədəf saytları üçün fərqli bir yaxınlıq göstərir[5].
Tarix və əsas eksperimental texnika[redaktə | mənbəni redaktə et]
Bir çox transkripsiya faktorları, məhdudlaşdırıcı fermentlər və sahəyə xas rekombinazlar üçün DNT-yə bağlanma sahələri müxtəlif eksperimental metodlardan istifadə edilərək kəşf edilmişdir[5]. Tarixən, DNT bağlanma yerlərinin aşkarlanması və təhlili üçün üstünlük verilən eksperimental metodlar analizi və elektroforetik hərəkət dəyişmə analizi (EMSA) olmuşdur. Bununla birlikdə, DNT mikro şüalarının və sürətli ardıcıllıq metodlarının inkişafı, bağlama sahələrini təyin etmək üçün yeni, kütləvi paralel üsullara. Xüsusi DNT bağlanma yerləri olan zülalların və digər molekulların bağlanma yaxınlığını ölçmək üçün biofiziki metod Mikroskale Termoporesis istifadə olunur[11].
Verilənlər bazası[redaktə | mənbəni redaktə et]
Bağlayıcı yerləri müəyyənləşdirmək üçün istifadə olunan müxtəlif eksperimental metodlar və əksər orqanizmlərin heterogen əhatə dairəsi və transkripsiya amillərinə görə DNT-nin bağlanma sahələri üçün mərkəzi məlumat bazası (Milli Biotexnologiya Məlumat Mərkəzindəki GenBank-a bənzər) mövcud deyil[12] . NCBI, nəzarət ardıcıllığında (RefSeq) DNT bağlanma sahəsinin şərhini götürməsinə baxmayaraq, bu məlumatlar təqdimatların əksəriyyətində mövcud deyil[13] . Bundan əlavə, bioinformatikanın DNT-yə bağlanma sahələrini proqnozlaşdırmaq üçün səmərəli alətlər yaratmaqda məhdud müvəffəqiyyəti (böyük yanlış pozitivlər tez-tez siliko motifinin aşkarlanması sahə tapmaq metodları ilə əlaqələndirilir) səbəbindən kompüter dəstəkli izahat vermək üçün sistematik bir səy göstərilməmişdir[14].
DNT bağlanma yerlərinin nümayişi[redaktə | mənbəni redaktə et]
DNT molekulu biri köhnə, digəri yeni olmaqla 2 zəncirdən ibarətdir. Bu proses reduplikasiya (ikiləşmə) adlanır. DNT sintezi prosesi bütövlükdə fermentlər qrupunun iştirakı ilə gedir[15]. DNT molekulunun cütləşməsi çox dəqiqliklə baş verir, yeni molekul əvvəlki molekulun tam oxşarı olur . Bəzi kimyəvi və fiziki faktorlar (ultrabənövşəyi və ionlaşdırıcı şülar, yüksək tempratur və s.) sintez olunan DNT zəncirinin quruluşunda dəyişikliklər yarada bilər . Molekulda belə zədələnmə yarandıqda, bu, adətən xüsusi ferment tərəfindən ləğv edilir və onun tamlığı bərpa olunur[16]. Ümumiyyətlə bir DNT bağlama motivi olaraq adlandırılan bir sıra DNT bağlama yerləri bir konsensus ardıcıllığı ilə təmsil edilə bilər. Bu baxış kompakt olmağın üstünlüyünə malikdir, lakin əhəmiyyətli miqdarda məlumatı görməməzlikdən gəlmək hesabına[17]. Bağlayıcı saytları təmsil etməyin daha dəqiq yolu, mövqeyə bağlı tezlik matrislərindən (PSFM) istifadə etməkdir. Bu matrislər, DNT ilə əlaqələndirici motivin hər mövqeyindəki hər bir bazanın tezliyi barədə məlumat verir[18]. . PSFM-lər ümumiyyətlə gizli mövqe fərziyyəsini qəbul edirlər (DNT bağlanma sahəsindəki fərqli mövqelər saytın fəaliyyətinə müstəqil şəkildə kömək edir), baxmayaraq ki, bəzi DNT bağlanma sahələri üçün bu fərziyyə mübahisəlidir. PSFM-dəki tezlik məlumatları, məlumat nəzəriyyəsi çərçivəsində rəsmi olaraq şərh edilə bilər , bunun ardıcıllıq loqosu şəklində qrafik şəkildə təmsil olunmasına gətirib çıxarır[19] .
Hesablama Axtarışı[redaktə | mənbəni redaktə et]
Bioinformatikada DNT-yə bağlanma sahələri ilə bağlı iki ayrı problem ayırd edilə bilər:
- bilinən DNT bağlayıcı motifin əlavə üzvlərinin axtarışı (sahə tapmaq problemi).
- funksional olaraq əlaqəli ardıcıllıqlar kolleksiyalarında yeni DNT bağlayan motivlərin kəşfi (bir sıra motivini tapmaq problemi). Onların əksəriyyəti məlumat nəzəriyyəsi prinsiplərinə güvənir və veb serverləri (Yellaboina) (Munch) mövcuddur, digərləri isə süni sinir şəbəkələri kimi maşın öyrənmə üsullarına müraciət edirlər. Ardıcıllıq motivlərini aşkar etmək üçün bir çox alqoritm də mövcuddur[20]. Bu metodlar bir sıra ardıcıllığın funksional səbəblərə görə ümumi bir bağlama motivinə malik olduğu fərziyyəsinə əsaslanır[21]. Məcburi motivlərin aşkarlanması metodları şərti olaraq sadalanan, deterministik və stoxastik olaraq bölünə bilər. DNT bağlanma motivlərini aşkarlamaq üçün tamamilə stokastik bir metodun yayılmış bir tətbiqidir.[22]. Bağlayıcı yerləri tapmaq və açma motivlərini tapmaq üçün daha mürəkkəb metodlar baza qatlanması və DNT bazaları arasındakı digər qarşılıqlı əlaqələrə əsaslanır, lakin DNT-də bağlanma yerləri üçün ümumiyyətlə mövcud olan kiçik nümunə ölçüləri səbəbindən, effektivliyi hələ tam olaraq istifadə edilməyib[23][24].
İstinadlar[redaktə | mənbəni redaktə et]
- ↑ Halford E.S.; Marko J.F. "How do site-specific DNA-binding proteins find their targets?". Nucleic Acids Research. 32 (10). 2004: 3040–3052. doi:10.1093/nar/gkh624. PMC 434431. PMID 15178741.
- ↑ Borneman, A.R.; Gianoulis, T.A.; Zhang, Z.D.; Yu, H.; Rozowsky, J.; Seringhaus, M.R.; Wang, L.Y.; Gerstein, M.; Snyder, M. "Divergence of transcription factor binding sites across related yeast species". Science. 317 (5839). 2007: 815–819. Bibcode:2007Sci...317..815B. doi:10.1126/science.1140748. PMID 17690298.
- ↑ Stormo GD. "DNA binding sites: representation and discovery". Bioinformatics. 16 (1). 2000: 16–23. doi:10.1093/bioinformatics/16.1.16. PMID 10812473.
- ↑ Pingoud A, Jeltsch A. "Recognition and Cleavage of DNA by Type-II Restriction Endonucleases". European Journal of Biochemistry. 246 (1). 1997: 1–22. doi:10.1111/j.1432-1033.1997.t01-6-00001.x. PMID 9210460.
- ↑ 5,0 5,1 5,2 Nash H. A. "Integrative recombination of bacteriophage lambda DNA in vitro". Proceedings of the National Academy of Sciences of the United States of America. 72 (3). 1975: 1072–1076. Bibcode:1975PNAS...72.1072N. doi:10.1073/pnas.72.3.1072. PMC 432468. PMID 1055366.
- ↑ Gyohda A, Komano T. "Purification and characterization of the R64 shufflon-specific recombinase". Journal of Bacteriology. 182 (10). 2000: 2787–2792. doi:10.1128/JB.182.10.2787-2792.2000. PMC 101987. PMID 10781547.
- ↑ Birge, E.A. 15: Site Specific Recombination // Bacterial and Bacteriophage Genetics (5th). Springer. 2006. 463–478. ISBN 978-0-387-23919-4.
- ↑ 8,0 8,1 Campbell A. "Fine Structure Genetics and its Relation to Function". Annual Review of Microbiology. 17 (1). 1963: 2787–2792. doi:10.1146/annurev.mi.17.100163.000405. PMID 14145311.
- ↑ Gilbert W, Maxam A. "The nucleotide sequence of the lac operator". Proceedings of the National Academy of Sciences of the United States of America. 70 (12). 1973: 3581–3584. Bibcode:1973PNAS...70.3581G. doi:10.1073/pnas.70.12.3581. PMC 427284. PMID 4587255.
- ↑ Maniatis T, Ptashne M, Barrell BG, Donelson J. "Sequence of a repressor-binding site in the DNA of bacteriophage lambda". Nature. 250 (465). 1974: 394–397. Bibcode:1974Natur.250..394M. doi:10.1038/250394a0. PMID 4854243.
- ↑ Elnitski L, Jin VX, Farnham PJ, Jones SJ. "Locating mammalian transcription factor binding sites: a survey of computational and experimental techniques". Genome Research. 16 (12). 2006: 1455–1464. doi:10.1101/gr.4140006. PMID 17053094.
- ↑ Baaske P, Wienken CJ, Reineck P, Duhr S, Braun D. "Optical Thermophoresis quantifies Buffer dependence of Aptamer Binding". Angew. Chem. Int. Ed. 49 (12). Feb 2010: 2238–41. doi:10.1002/anie.200903998. PMID 20186894. Lay summary – Phsyorg.com.
- ↑ Wienken CJ; və b. "Protein-binding assays in biological liquids using microscale thermophoresis". Nature Communications. 1 (7). 2010: 100. Bibcode:2010NatCo...1..100W. doi:10.1038/ncomms1093. PMID 20981028.
- ↑ Schneider T.D. "Consensus sequence Zen". Applied Bioinformatics. 1 (3). 2002: 111–119. PMC 1852464. PMID 15130839.
- ↑ Bulyk M.L.; Johnson P.L.; Church G.M. "Nucleotides of transcription factor binding sites exert interdependent effects on the binding affinities of transcription factors". Nucleic Acids Research. 30 (5). 2002: 1255–1261. doi:10.1093/nar/30.5.1255. PMC 101241. PMID 11861919.
- ↑ Schneider TD, Stormo GD, Gold L, Ehrenfeucht A. "Information content of binding sites on nucleotide sequences". Journal of Molecular Biology. 188 (3). 1986: 415–431X. doi:10.1016/0022-2836(86)90165-8. PMID 3525846.
- ↑ Ganji, Mahipal; Docter, Margreet; Le Grice, Stuart F. J.; Abbondanzieri, Elio A. "DNA binding proteins explore multiple local configurations during docking via rapid rebinding". Nucleic Acids Research. 44 (17). 2016-09-30: 8376–8384. doi:10.1093/nar/gkw666. ISSN 0305-1048. PMC 5041478. PMID 27471033.
- ↑ Erill I; O'Neill MC. "A reexamination of information theory-based methods for DNA-binding site identification". BMC Bioinformatics. 10 (1). 2009: 57. doi:10.1186/1471-2105-10-57. PMC 2680408. PMID 19210776.
- ↑ O'Neill M.C. "Training back-propagation neural networks to define and detect DNA-binding sites". Nucleic Acids Research. 19 (2). 1991: 133–318. doi:10.1093/nar/19.2.313. PMC 333596. PMID 2014171.
- ↑ Bailey T.L. Discovering sequence motifs // Bioinformatics (PDF). Methods in Molecular Biology. Methods in Molecular Biology™. 452. 2008. 231–251. doi:10.1007/978-1-60327-159-2_12. ISBN 978-1-58829-707-5. PMID 18566768.
- ↑ Bailey T.L. "Discovering novel sequence motifs with MEME". Current Protocols in Bioinformatics. 2 (4). 2002: 2.4.1–2.4.35. doi:10.1002/0471250953.bi0204s00. PMID 18792935.
- ↑ Das MK, Dai HK. "A survey of DNA motif finding algorithms". BMC Bioinformatics. 8 (Suppl 7). 2007: S21. doi:10.1186/1471-2105-8-S7-S21. PMC 2099490. PMID 18047721.
- ↑ Siddharthan R, Siggia ED, van Nimwegen E. "PhyloGibbs: A Gibbs sampling motif finder that incorporates phylogeny". PLOS Comput Biol. 1 (7). 2005: e67. Bibcode:2005PLSCB...1...67S. doi:10.1371/journal.pcbi.0010067. PMC 1309704. PMID 16477324.
- ↑ Salama RA, Stekel DJ. "Inclusion of neighboring base interdependencies substantially improves genome-wide prokaryotic transcription factor binding site prediction". Nucleic Acids Research. 38 (12). 2010: e135. doi:10.1093/nar/gkq274. PMC 2896541. PMID 20439311.